
66
Notas de Investigação
A malária é uma doença devastadora, transmitida
por vetores, que reivindica quase meio milhão de
vidas humanas a cada ano (WHO, 2017). A grande
maioria das mortes é causada por
Plasmodium falci-
parum
, a espécie mais letal de parasitas de malária
humana. A introdução de derivados de artemisinina
combinados com fármacos quimicamente distintos
sob uma prática conhecida como Terapia Combinada
com Artemisinina (ACT), para o tratamento de in-
feções por
P. falciparum
, tem sido extre-
mamente bem sucedida na redução do
número de casos e óbitos relacionados
à malária nos últimos 15 anos. No en-
tanto, à semelhança de todos ou outros
antimaláricos, o parasita acabou tam-
bém por evoluir resistência a estes fár-
macos (Haldar et al, 2018; Figura 1).
Deste modo, a identificação dos ge-
nes subjacentes à resistência assume-
-se como uma prioridade fundamental.
No entanto, o conhecimento atual so-
bre mecanismos de resistência a drogas
antimaláricas é limitado pelo fato de
a grande parte das abordagens dispo-
níveis se concentrarem na procura de
mutações em um número pequeno de
genes. Assim, o objetivo do nosso mo-
delo de trabalho é investigar as caracte-
rísticas evolutivas, genéticas e genómicas determi-
nantes de quimioresistência em parasitas de malária,
utilizando uma abordagem que permite explorar a
globalidade do genoma do Plasmódio, detetando to-
das as mutações responsáveis pela resistência. Este
paradigma faz uso de uma linhagem de
Plasmodium
chabaudi
isogénica, denominada AS, constituída por
parasitas mutantes resistentes a diversas drogas, tais
como cloroquina, mefloquina e derivados da artemi-
sinina (Tabela 1).
Estes parasitas fo-
ram utilizados para
uma rápida deteção
de
loci
associados a
diferentes
fenóti-
pos, à escala genética
global, usando uma
tecnologia denomi-
nada
Linkage Group
Selection
(Carter et al,
2007; Figura 2). Em
complemento, os ge-
nomas completos dos
parasitas resistentes
foram sequenciados
e comparados com
os dos seus progeni-
tores sensíveis, usan-
do tecnologias de
sequenciamento de
genomas de segunda
geração (Solexa, Illu-
mina; Figura 3).
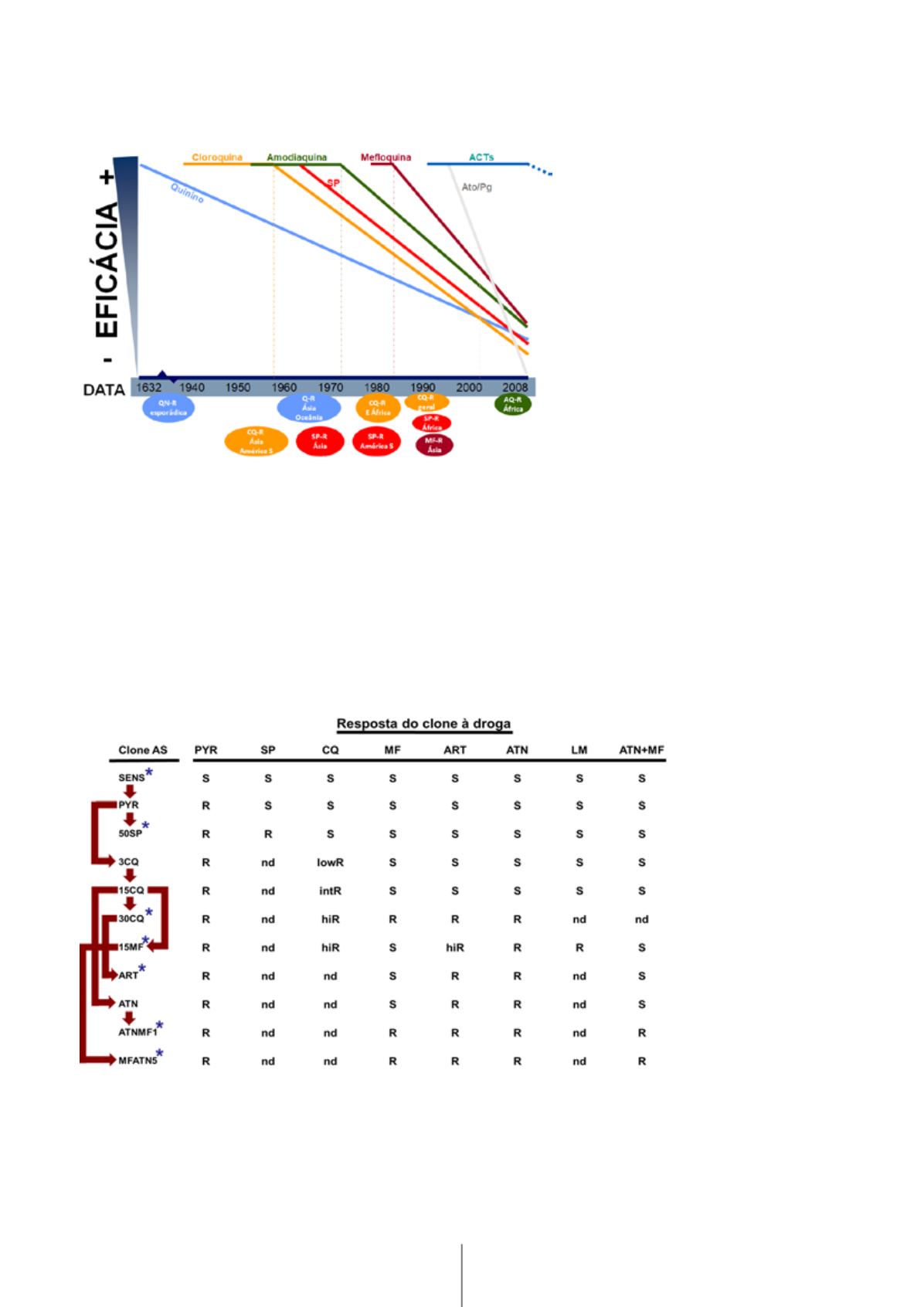
Tabela 1.
LinhagemAS de
P. chabaudi
A denominação dos clones de parasitas é apresentada na primeira coluna da esquerda. As restantes colunas da figura
representam o fenótipo de cada clone à droga em questão. PYR: pirimetamina; SP: sulfadoxina + pirimetamina; CQ:
cloroquina; MF: mefloquina; ART: artemisinina; ATN: artesunato; LM: lumefantrina; S: sensível; R: resistente; lowR:
baixa resistência; intR: resistência intermédia; nd: não determinado. Todos os clones resistentes foram derivados do seu
progenitor sensível através de selecção
in vivo
por pressão continua de fármaco (setas bordeaux).Asteriscos denominam os
parasitas cujo genoma foi totalmente resequenciado. Fonte: autoria própria
Fig.1 -
Evolução cronológica da resistência ao diferentes fármacos antimaláricos em
P.falciparum
,
de acordo com área geográfica


















